Lär dig hur du använder OCR-verktyg, Apache Spark och andra Apache Hadoop-komponenter för att bearbeta PDF-bilder i skala.
Teknikerna för optisk teckenigenkänning (OCR) har utvecklats avsevärt under de senaste 20 åren. Men under den tiden har det inte gjorts några eller inga ansträngningar att kombinera OCR med distribuerade arkitekturer som Apache Hadoop för att bearbeta ett stort antal bilder i nästan realtid.
I det här inlägget kommer du att lära dig hur du använder standardverktyg med öppen källkod tillsammans med Hadoop-komponenter som Apache Spark, Apache Solr och Apache HBase för att göra just det för ett användningsfall för medicinsk utrustning. Specifikt kommer du att använda en offentlig datauppsättning för att konvertera berättande text till sökbara fält.
Även om det här exemplet koncentrerar sig på information om medicintekniska produkter, kan det användas i många andra scenarier där bearbetning och beständiga bilder krävs. Försäkringsbolag kan till exempel göra alla sina skannade dokument i skadefiler sökbara för bättre skadelösning. På samma sätt kan leveranskedjan i en tillverkningsanläggning skanna alla tekniska datablad från reservdelsleverantörer och göra dem sökbara av analytiker.
Användningsfall:Registrering av medicinsk utrustning
De senaste åren har sett en uppsjö av förändringar inom området elektronisk läkemedelsproduktregistrering. IDMP (Identifiering av medicinska produkter) ISO-standard är ett sådant meddelandeformat för registrering av produkter och substanserna i dem, där läkemedels-ID, förpacknings-ID och batch-ID används för att spåra produkterna i fall av negativa upplevelser, olagligt import, förfalskning och andra frågor om säkerhetsövervakning. Standarden kräver att inte bara nya produkter måste registreras, utan att den äldre/arkiverade arkiveringen av varje produkt som allmänheten kan utsättas för också måste tillhandahållas i elektronisk form.
För att följa IDMP-standarder i olika företag måste företag kunna hämta och bearbeta data från flera datakällor, såsom RDBMS samt, i vissa fall, äldre produktdatablad. Även om det är välkänt hur man matar in data från RDBMS via tekniker som Apache Sqoop, kräver äldre dokumentbehandling lite mer arbete. För det mesta måste dokumenten matas in och relevant text måste extraheras programmatiskt i skala med hjälp av befintlig OCR-teknik.
Datauppsättning
Vi kommer att använda en datamängd från FDA som innehåller alla 510(k)-ansökningar som någonsin lämnats in av tillverkare av medicintekniska produkter sedan 1976. Section 510(k) i Food, Drug and Cosmetic Act kräver att enhetstillverkare som måste registrera sig ska meddela FDA om deras avsikt att marknadsföra en medicinsk utrustning minst 90 dagar i förväg.
Denna datauppsättning är användbar av flera skäl i detta fall:
- Datan är gratis och allmän egendom.
- Datan passar in i den europeiska förordningen, som aktiveras i juli 2016 (där tillverkare måste följa nya datastandarder). FDA-fyllningar har viktig information som är relevant för att få en fullständig bild av IDMP.
- Formatet på dokumenten (PDF) gör att vi kan demonstrera enkla men effektiva OCR-tekniker när vi hanterar dokument i flera format.
För att effektivt indexera dessa data måste vi extrahera några fält från bilderna. Nedan är ett exempeldokument med de potentiella fälten som kan extraheras.
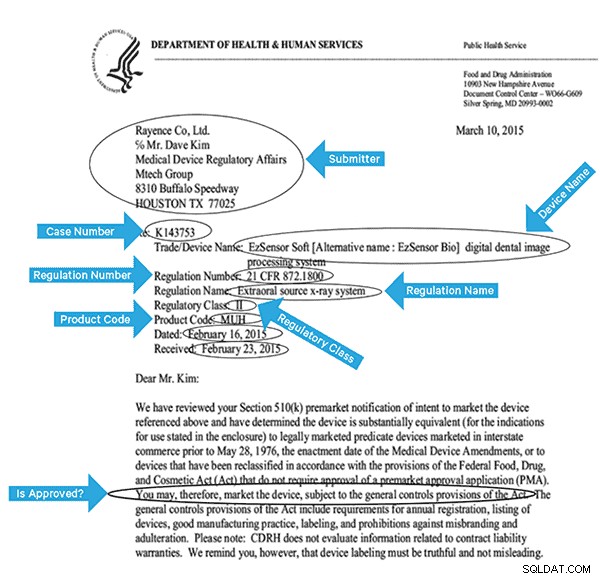
Högnivåarkitektur
För detta användningsfall lagras PDF-filerna i HDFS och bearbetas med Spark- och OCR-bibliotek. (Inmatningssteget ligger utanför omfattningen av det här inlägget, men det kan vara så enkelt som att köra hdfs -dfs -put eller med ett webhdfs-gränssnitt.) Spark tillåter användning av nästan identisk kod i en Spark Streaming-applikation för streaming i nästan realtid, och HBase är ett perfekt lagringsmedium för slumpmässig åtkomst med låg latens—och är väl lämpat för lagring av bilder, med den nya MOB-funktionen, för att starta. Cloudera Search (som är byggt ovanpå Apache Solr) är den enda söklösningen som integreras med HBase, vilket gör att du kan bygga sekundära index.
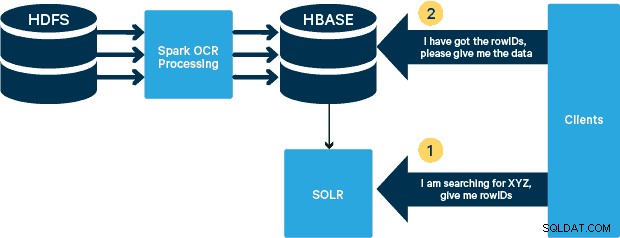
Konfigurera tabellen för medicinska enheter i HBase
Vi kommer att hålla schemat för vårt användningsfall enkelt. Rad-ID kommer att vara filnamnet, och det kommer att finnas två kolumnfamiljer:"info" och "obj". Kolumnfamiljen "info" kommer att innehålla alla fält som vi extraherat från bilderna. Kolumnfamiljen "obj" kommer att innehålla byten för det faktiska binära objektet, i detta fall PDF. Namnet på tabellen i vårt fall kommer att vara "mdds."
Vi kommer att dra fördel av HBase MOB (medium object) funktionalitet som introduceras i HBASE-11339. För att ställa in HBase för att hantera MOB krävs några extra steg, men bekvämt kan du hitta instruktioner på den här länken.
Det finns många sätt att skapa tabellen i HBase programmatiskt (Java API, REST API eller liknande metod). Här kommer vi att använda HBase-skalet för att skapa "mdds"-tabellen (avsiktligt med hjälp av ett beskrivande kolumnfamiljenamn för att göra saker lättare att följa). Vi vill ha "info"-kolumnfamiljen replikerad till Solr, men inte MOB-data.
Kommandot nedan kommer att skapa tabellen och aktivera replikering på en kolumnfamilj som heter "info." Det är viktigt att ange alternativet REPLICATION_SCOPE => '1' , annars kommer HBase Lily Indexer inte att få några uppdateringar från HBase. Vi vill använda MOB-sökvägen i HBase för objekt större än 10MB. För att åstadkomma det skapar vi också en annan kolumnfamilj, kallad "obj", med hjälp av följande parametrar för MOB:er:
IS_MOB => sant, MOB_THRESHOLD => 10240000
IS_MOB parametern anger om denna kolumnfamilj kan lagra MOB, medan MOB_THRESHOLD anger efter hur stort objektet måste vara för att det ska betraktas som en MOB. Så låt oss skapa tabellen:
skapa 'mdds', {NAME => 'info', DATA_BLOCK_ENCODING => 'FAST_DIFF',REPLICATION_SCOPE => '1'},{NAME => 'obj', IS_MOB => true, MOB_THRESHOLD => 10240000} För att bekräfta att tabellen skapades korrekt, kör följande kommando i HBase-skalet:
hbase(main):001:0> beskriv 'mdds'Tabell mdds är ENABLEDmddsCOLUMN FAMILIES DESCRIPTION{NAME => 'info', DATA_BLOCK_ENCODING => 'FAST_DIFF', BLOOMFILTER => 'RAD', => '1CO' , VERSIONS => '1', KOMPRESSION => 'INGEN', MIN_VERSIONS => '0', TTL => 'FOR ALLTID', KEEP_DELETED_CELLS => 'FALSK', BLOCKSIZE => '65536', IN_MEMORY => 'falskt', BLOCKCACHE => 'true'}{NAME => 'obj', DATA_BLOCK_ENCODING => 'INGEN', BLOOMFILTER => 'RAD', REPLICATION_SCOPE => '0', KOMPRESSION => 'INGEN', VERSIONER => '1', MIN_VERSIONS => '0', TTL => 'FÖR ALLTID', MOB_THRESHOLD => '10240000', IS_MOB => 'sant', KEEP_DELETED_CELLS => 'FALSK', BLOCKSIZE => '65536', IN_MEMORY' => 'falskt' => 'true'}2 rad(er) på 0,3440 sekunder Bearbeta skannade bilder med Tesseract
OCR har kommit långt när det gäller att hantera teckensnittsvariationer, bildbrus och justeringar. Här kommer vi att använda OCR-motorn med öppen källkod Tesseract, som ursprungligen utvecklades som proprietär programvara vid HP-labb. Tesseract-utveckling har sedan dess släppts som en programvara med öppen källkod och sponsrats av Google sedan 2006.
Tesseract är ett mycket portabelt mjukvarubibliotek. Den använder Leptonicas bildbehandlingsbibliotek för att generera en binär bild genom att göra adaptiv tröskelvärde på en grå eller färgad bild.
Bearbetningen följer en traditionell steg-för-steg pipeline. Följande är det grova flödet av steg:
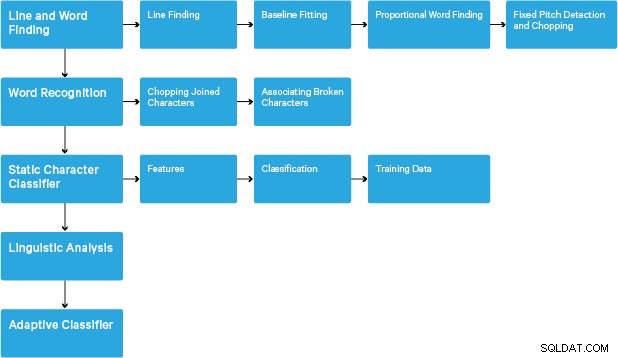
Bearbetningen börjar med en ansluten komponentanalys, vilket resulterar i att de hittade komponenterna lagras. Det här steget hjälper till vid inspektion av kapsling av konturer och antalet barn- och barnbarnskonturer.
I detta skede samlas konturerna samman, enbart genom kapsling, till binära stora objekt (BLOBs). BLOB är organiserade i textrader och linjerna och områdena analyseras för fast tonhöjd eller proportionell text. Textrader delas upp i ord på olika sätt beroende på typ av teckenavstånd. Text med fast tonhöjd hackas omedelbart av teckenceller. Proportionell text delas upp i ord med bestämda mellanslag och otydliga mellanslag.
Igenkännandet fortsätter sedan som en tvåstegsprocess. I första passet görs ett försök att känna igen varje ord i tur och ordning. Varje ord som är tillfredsställande skickas till en adaptiv klassificerare som träningsdata. Den adaptiva klassificeraren får då en chans att mer exakt känna igen text längre ner på sidan. Eftersom den adaptiva klassificeraren kan ha lärt sig något användbart för sent för att göra ett bidrag nära toppen av sidan, körs ett andra pass över sidan, där ord som inte kändes igen tillräckligt väl igenkänns igen. En sista fas löser suddiga mellanslag och kontrollerar alternativa hypoteser för x-höjden för att hitta text med liten bokstäver.
Tesseract i sin nuvarande form är helt unicode-kapabel och tränad för flera språk. Baserat på vår forskning är det ett av de mest exakta biblioteken med öppen källkod som finns för OCR. Som tidigare nämnts använder Tesseract Leptonica. Vi använder oss också av Ghostscript för att dela upp PDF-filerna i bilder. (Du kan dela upp i bildkomprimeringsformat efter eget val; vi valde PNG.) Dessa tre bibliotek är skrivna i C++, och för att anropa dem från Java/Scala-program måste vi använda implementeringar av motsvarande Java Native Interfaces. I vårt arbete använder vi JNI-bindningarna från JavaPresets. (Bygginstruktionerna finns nedan.) Vi använde Scala för att skriva Spark-drivrutinen.
val renderer :SimpleRenderer =ny SimpleRenderer( )renderer.setResolution( 300 )val images:List[Image] =renderer.render( document )
Leptonica läser in de delade bilderna från föregående steg.
ImageIO.write( x.asInstanceOf[RenderedImage], "png", imageByteStream)val pix:PIX =pixReadMem ( ByteBuffer.wrap( imageByteStream.toByteArray( ) ).array( ), ByteBuffer.StrapAram(rayByteByteAram) ) ).capacity( ))
Vi använder sedan Tesseract API-anrop för att extrahera texten. Vi antar att dokumenten är på engelska här, därför är den andra parametern till Init-metoden "eng."
val api:TessBaseAPI =new TessBaseAPI( )api.Init( null, "eng" )api.SetImage(pix)api.GetUTF8Text().getString()
Efter att bilderna har bearbetats extraherar vi några fält från texten och skickar dem till HBase.
def populateHbase ( fileName:String, lines:String, pdf:org.apache.spark.input.PortableDataStream) :Unit ={ /** Konfigurera och öppna en HBase-anslutning */ val mddsTbl =_conn.getTable( TableName. valueOf( "mdds" )); val cf ="info" val put =new Put( Bytes.toBytes( filnamn )) /** * Extrahera fält här med hjälp av Regexes * Skapa Put-objekt och skicka till HBase */ val aAndCP ="""(?s)(? m).*\d\d\d\d\d-\d\d\d\d(.*)\nRe:(\w\d\d\d\d\d\d).*"" ".r …….. rader matchar { case aAndCP( addr, casenum ) => put.add( Bytes.toBytes( cf ),Bytes.toBytes( "submitter_info" ),Bytes.toBytes( adr ) ).add( Bytes .toBytes( cf ),Bytes.toBytes( "case_num" ), Bytes.toBytes( casenum )) case _ => println( "matchade inte ett regex") } ……. lines.split("\n").foreach { val regNumRegex ="""Föreskriftsnummer:\s+(.+)""".r val regNameRegex ="""Föreskriftsnamn:\s+(.+)""" .r …….. ……. _ matcha { case regNumRegex( regNum ) => put.add( Bytes.toBytes( cf ),Bytes.toBytes( "reg_num" ), ……. ….. case _ => print( "" ) } } put.add ( Bytes.toBytes( cf ), Bytes.toBytes( "text" ), Bytes.toBytes( lines )) val pdfBytes =pdf.toArray.clone put.add(Bytes.toBytes( "obj" ), Bytes.toBytes( " pdf" ), pdfBytes ) mddsTbl.put( put ) …….} Om du tittar noga på koden ovan, precis innan vi skickar Put-objektet till HBase, infogar vi de råa PDF-bytena i "obj"-kolumnfamiljen i tabellen. Vi använder HBase som lagringslager för de extraherade fälten samt råbilden. Detta gör det snabbt och bekvämt för applikationen att extrahera originalbilden, om det behövs. Hela koden finns här. (Det är värt att notera att även om vi använde standard HBase API:er för att skapa Put-objekt för HBase, i ett riktigt produktionssystem, skulle det vara klokt att överväga att använda SparkOnHBase API:er, som möjliggör batchuppdateringar av HBase från Spark RDDs.)
Execution Pipeline
Vi kunde bearbeta varje PDF i ett seriellt ramverk. För att skala bearbetningen valde vi att bearbeta dessa PDF-filer på ett distribuerat sätt med hjälp av Spark. Följande diagram visar hur vi kombinerar olika stadier av denna bearbetning för att förvandla arbetsflödet till ett enkelt makroanrop från Spark och få data att laddas in i HBase.
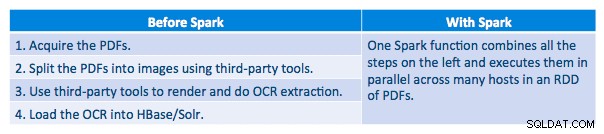
Vi försökte också göra en jämförelse mellan serialiseringsmetoder, men med vår datauppsättning såg vi ingen signifikant skillnad i prestanda.
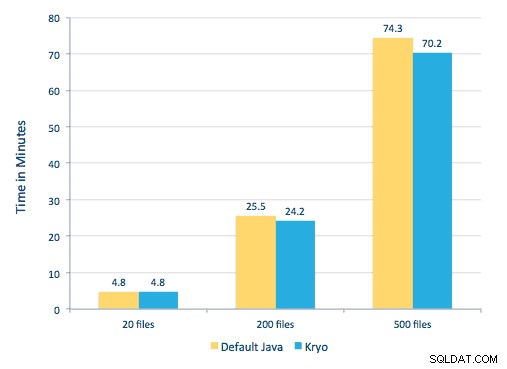
Miljöinställningar
Använd maskinvara:Fem-nodskluster med 15 GB minne, 4 vCPU:er och 2x40 GB SSD
Eftersom vi använde C++-bibliotek för bearbetning använde vi JNI-bindningarna som finns här.
Bygg JNI-bindningarna för Tesseract och Leptonica från javaCPP-förinställningar:
-
- På alla noder:
yum -y install automake autoconf libtool zlib-devel libjpeg-devel giflib libtiff-devel libwebp libwebp-devel libicu-devel openjpeg-devel cairo-devel git clone https://github.com/bytedeco/javacpp-presets.gitcd javacpp-presets- Bygg Leptonica.
cd leptonica./cppbuild.sh installera leptonicacd cppbuild/linux-x86_64/leptonica-1.72/LDFLAGS="-Wl,-rpath -Wl,/usr/local/lib" ./configuremake &&sudo make installcd ../../../mvn clean installcd ..
- Bygg Tesseract.
- På alla noder:
cd tesseract./cppbuild.sh installera tesseractcd tesseract/cppbuild/linux-x86_64/tesseract-3.03LDFLAGS="-Wl,-rpath -Wl,/usr/local/lib" ./configuremake &&make installcd ../ ../../mvn clean installcd ..
- Bygg javaCPP-förinställningar.
mvn clean install --projects leptonica,tesseract
Vi använder Ghostscript för att extrahera bilderna från PDF-filerna. Instruktioner för att bygga Ghostscript, som motsvarar versionerna av Tesseract och Leptonica som används här, är följande. (Se till att Ghostscript inte är installerat i systemet via pakethanteraren.)
wget https://downloads.ghostscript.com/public/ghostscript-9.16.tar.gztar zxvf ghostscript-9.16.tar.gzcd ghostscript-9.16./autogen.sh &&./configure --prefix=/usr - -disable-compile-inits --enable-dynamicsudo gör &&gör såinstallera &&installera -v -m644 base/*.h /usr/include/ghostscript &&ln -v -s ghostscript /usr/include/ps(Beroende på din ldpath inställning kan du behöva göra):sudo ln -sf /usr/lib/libgs.so /usr/local/lib/libgs.so
Se till att alla nödvändiga bibliotek finns i klassvägen. Vi lägger alla relevanta burkar i en katalog som heter lib. Komma är viktigt nedan:
$ för i i `ls lib/*`; exportera MY_JARS=./$i,$MY_JARS; donetesseract.jar, tesseract-linux-x86_64.jar, javacpp.jar, ghost4j-1.0.0.jar, leptonica.jar, leptonica-1.72-1.0.jar, leptonica-linux-x86_64.jar
Vi åberopar Spark-programmet enligt följande. Vi måste specificera extraLibraryPath för infödda Ghostscript-bibliotek; den andra konf. behövs för Tesseract.
spark-submit --jars $MY_JARS --num-executors 12 --executor-memory 4G --executor-cores 1 --conf spark.executor.extraLibraryPath=/usr/local/lib --confspark.executorEnv. TESSDATA_PREFIX=/home/vsingh/javacpp-presets/tesseract/cppbuild/1-x86_64/share/tessdata/ --confspark.executor.extraClassPath=/etc/hbase/conf:/opt/cloudera/parcels/CDH/lib/hbase /lib/htrace-core-3.1.0-incubating.jar --driver-class-path/etc/hbase/conf:/opt/cloudera/parcels/CDH/lib/hbase/lib/htrace-core-3.1.0 -incubating.jar --conf spark.serializer=org.apache.spark.serializer.KryoSerializer--conf spark.kryoserializer.buffer.mb=24 --class com.cloudera.sa.OCR.IdmpExtraction
Skapa en Solr-samling
Solr integreras ganska sömlöst med HBase via Lily HBase Indexer. För att förstå hur integrationen av Lily Indexer-integrering med HBase går till kan du fräscha upp via vårt tidigare inlägg i avsnittet "Förstå HBase-replikering och Lily HBase Indexer".
Nedan beskriver vi de steg som måste utföras för att skapa indexen:
- Generera ett exempel på schema.xml-konfigurationsfil:
solrctl --zk localhost:2181 instancedir --generate $HOME/solrcfg - Redigera filen schema.xml i
$HOME/solrcfg, och anger de fält vi behöver för vår samling. Hela filen finns här. - Ladda upp Solr-konfigurationerna till ZooKeeper:
solrctl --zk localhost:2181/solr instancedir --create mdds_collection $HOME/solrcfg - Generera Solr-samlingen med 2 skärvor (-s 2) och 2 repliker (-r 2):
solrctl --zk localhost:2181/solr --solr localhost:8983/solr collection --create mdds_collection -s 2 -r 2
I kommandot ovan skapade vi en Solr-samling med två shards (-s 2) och två replicas (-r 2) parametrar. Parametrarna var tillräckliga för vår korpus, men i en faktisk utbyggnad skulle man behöva ställa in siffran baserat på andra överväganden utanför vårt diskussionsområde här.
Registrera indexeraren
Detta steg behövs för att lägga till och konfigurera indexeraren och HBase-replikeringen. Kommandot nedan kommer att uppdatera ZooKeeper och lägga till mdds_indexer som en replikeringspeer för HBase. Den kommer också att infoga konfigurationer i ZooKeeper, som Lily HBase Indexer kommer att använda för att peka på rätt samling i Solr. |
hbase-indexer add-indexer -n mdds_indexer -c indexer-config.xml -cp solr.zk=localhost:2181/solr -cp solr.collection=mdds_collection.
Argument:
-n mdds_indexer– anger namnet på indexeraren som kommer att registreras i ZooKeeper-c indexer-config.xml– konfigurationsfil som kommer att specificera indexeringsbeteende-cp solr.zk=localhost:2181/solr– anger platsen för ZooKeeper och Solr config. Detta bör uppdateras med den miljöspecifika platsen för ZooKeeper.-cp solr.collection=mdds_collection– anger vilken samling som ska uppdateras. Kom ihåg Solr-konfigurationssteget där vi skapade samling1.
index-config.xml filen är relativt okomplicerad i det här fallet; allt det gör är att specificera för indexeraren vilken tabell som ska titta på, klassen som kommer att användas som mappare (com.ngdata.hbaseindexer.morphline.MorphlineResultToSolrMapper ), och platsen för Morphline-konfigurationsfilen. Som standard är mappningstyp inställd på rad , i vilket fall Solr-dokumentet blir hela raden. Param name="morphlineFile" anger platsen för Morphlines-konfigurationsfilen. Platsen kan vara en absolut sökväg till din Morphlines-fil, men eftersom du använder Cloudera Manager, ange den relativa sökvägen som morphlines.conf.
Innehållet i hbase-indexer-konfigurationsfilen kan hittas här.
Konfigurera och starta Lily HBase Indexer
När du aktiverar Lily HBase Indexer måste du ange Morphlines-transformationslogiken som gör att denna indexerare kan analysera uppdateringar till tabellen Medical Device och extrahera alla relevanta fält. Gå till Tjänster och välj Lily HBase Indexer som du lagt till tidigare. Välj Konfigurationer->Visa och redigera->Service-Wide->Morphlines . Kopiera och klistra in Morphlines-filen.
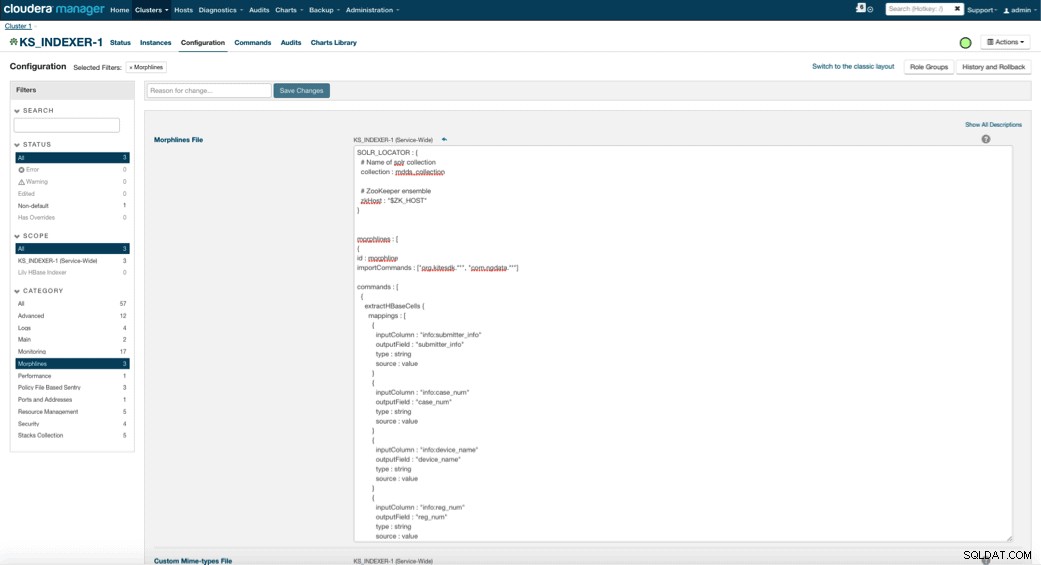
Biblioteket för medicinsk utrustning morphlines kommer att utföra följande åtgärder:
- Läs HBase-e-posthändelserna med
extractHBaseCellskommando - Konvertera datum-/tidsstämplarna till ett fält som Solr kommer att förstå, med
convertTimestampkommandon - Släpp alla extra fält som vi inte angav i schema.xml, med
sanitizeUknownSolrFieldskommando
Ladda ner en kopia av denna Morphlines-fil härifrån.
En viktig anmärkning är att id-fältet kommer att genereras automatiskt av Lily HBase Indexer. Den inställningen kan konfigureras i filen index-config.xml ovan genom att ange attributet unique-key-field. Det är en bästa praxis att lämna standardnamnet för id – eftersom det inte specificerades i xml-filen ovan genererades standard-id-fältet och kommer att vara en kombination av RowID.
Åtkomst till data
Du kan välja mellan många visuella verktyg för att komma åt de indexerade bilderna. HUE och Solr GUI är båda mycket bra alternativ. HBase möjliggör också ett antal åtkomsttekniker, inte bara från ett GUI utan också via HBase-skalet, API och till och med enkla skripttekniker.
Integration med Solr ger dig stor flexibilitet och kan även ge mycket enkla såväl som avancerade sökmöjligheter för din data. Att till exempel konfigurera Solr schema.xml-filen så att alla fält inom e-postobjektet lagras i Solr tillåter användare att få tillgång till fullständiga meddelandekroppar via en enkel sökning, med en avvägning av lagringsutrymme och beräkningskomplexitet. Alternativt kan du konfigurera Solr för att endast lagra ett begränsat antal fält, till exempel id. Med dessa element kan användare snabbt söka i Solr och hämta rowID som i sin tur kan användas för att hämta enskilda fält eller hela bilden från själva HBase.
Exemplet ovan lagrar endast rowID i Solr men indexerar på alla fält som extraherats från bilden. Genom att söka i Solr i det här scenariot hämtas HBase rad-ID, som du sedan kan använda för att fråga HBase. Den här typen av installation är idealisk för Solr eftersom den håller lagringskostnaderna låga och drar full nytta av Solrs indexeringsmöjligheter.
Exempel på frågor
Nedan finns några exempel på frågor som kan göras från applikationen till Solr. Tanken är att klienten initialt ska fråga Solr-index och returnera rowID från HBase. Fråga sedan HBase för resten av fälten och/eller den ursprungliga råbilden.
- Ge mig alla dokument som har lämnats in mellan följande datum:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=received:[2010-01 -06T23:59:59.999Z TILL 2010-02-06T23:59:59.999Z]
- Ge mig dokument vars arkiverades under regulatoriskt namn för mobila röntgensystem:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=reg_name:Mobile röntgensystem
- Ge mig alla dokument som har lämnats in från kinesiska tillverkare:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=submitter_info:*China*
ID:n från Solr-dokument är rad-ID:n i HBase; den andra delen av frågan kommer att gå till HBase för att extrahera data (inklusive den råa PDF-filen om det behövs).
Åtkomst via HUE
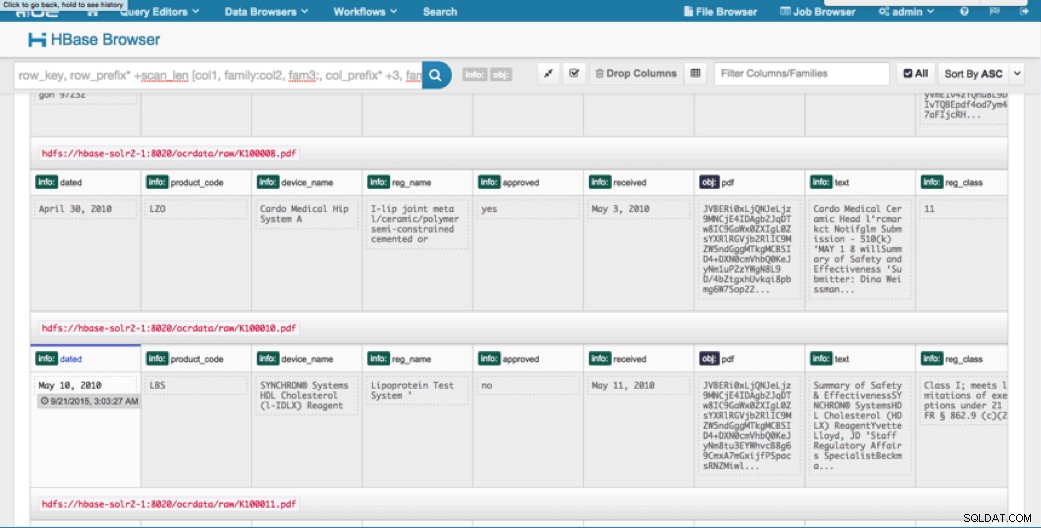
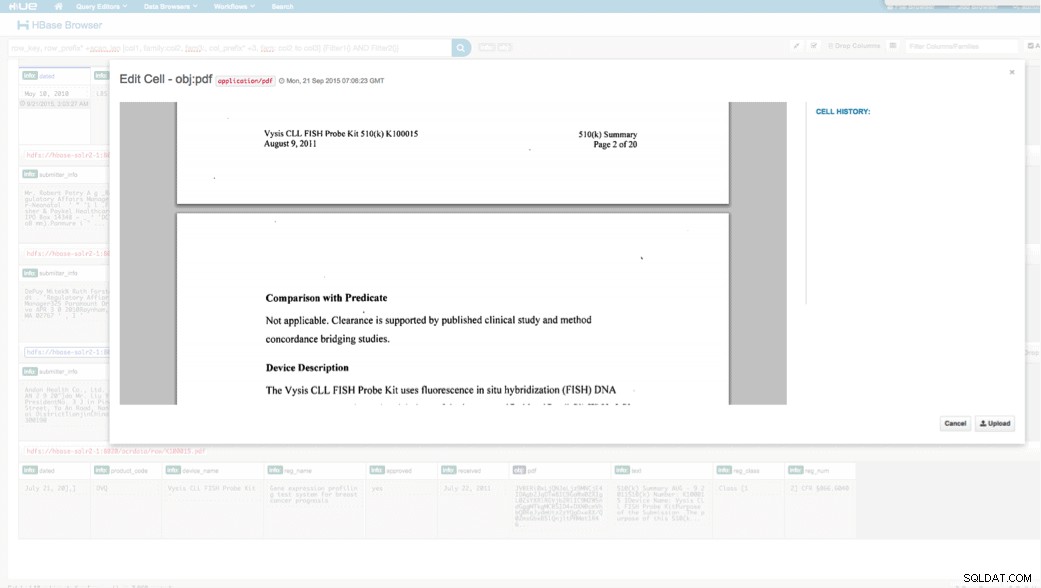
Vi kan se uppladdade data via HBase Browser i HUE. En bra sak med HUE är att den kan upptäcka binärfilerna för PDF och rendera dem när de klickas på.
Nedan är en ögonblicksbild av vyn av de analyserade fälten i HBase-rader och även en renderad vy av ett av PDF-objekten lagrade som en MOB i obj kolumnfamiljen.
Slutsats
I det här inlägget har vi demonstrerat hur man använder standardteknologier med öppen källkod för att utföra OCR på skannade dokument med hjälp av ett skalbart Spark-program, lagra i HBase för snabb hämtning och indexera den extraherade informationen i Solr. Det bör vara uppenbart att:
- Med tanke på meddelandespecifikationens format kan vi extrahera fält och värdepar och göra dem sökbara via Solr.
- Dessa fält från data kan uppfylla IDMP-kraven för att göra äldre data elektroniska, vilket träder i kraft någon gång nästa år.
- Fälten såväl som råbilder kan finnas kvar i HBase och nås via standard-API:er.
Om du känner att du behöver bearbeta skannade dokument och kombinera data med olika andra källor i ditt företag, överväg att använda en kombination av Spark, HBase, Solr, tillsammans med Tesseract och Leptonica. Det kan spara mycket tid och pengar!
Jeff Shmain är senior lösningsarkitekt på Cloudera. Han har 16+ års erfarenhet av finansbranschen med stor förståelse för säkerhetshandel, risker och regleringar. Under de senaste åren har han arbetat med olika användningsfallsimplementeringar hos 8 av 10 världens största investeringsbanker.
Vartika Singh är senior lösningskonsult på Cloudera. Hon har över 12 års erfarenhet av tillämpad maskininlärning och mjukvaruutveckling.
